Понятно, что в мозге животных, не исключая и человека, функционируют не сложные органические молекулы непосредственно, а организованные из них структуры более высокого порядка — нервные клетки, ткани и отделы, но всё же, всё же! В 1994 г. Леонард Адлеман (Leonard Adleman), информатик из Университета Южной Калифорнии в Лос-Анджелесе, опубликовал научную работу о созданном им «компьютере из ДНК-супа» — буквально заполненной водным раствором биомолекул пробирке. «Суп» оказался способен решать классическую задачу коммивояжёра, т. е. выявлять минимальный — по затраченному на его прохождение времени — маршрут, который позволит обойти конечное число точек, связанных заданным набором путевых линий. Для классических фон-неймановских машин весь класс подобных задач комбинаторной оптимизации — натуральная головная боль, поскольку они относятся к NP-полным, т. е. математиками до сих пор не обнаружено способа решать их за полиномиальное время. Да, эксперимент Адлемана при продолжительности собственно вычислительной сессии примерно в одну секунду (и всего для 7 точек) потребовал целой недели на подготовку, но смысл его был в практическом подтверждении самой возможности производить ДНК-вычисления с таким уровнем параллелизации, который полупроводниковым машинам на логике фон Неймана и не снился. Впрочем, вычисления на биомолекулярной элементной базе — тема для отдельного разговора: «компьютеры» такого рода страдают от сравнительно высокого уровня случайных ошибок, требуют разработки особых алгоритмов и подходят далеко не для каждого класса задач. А вот хранение данных на тех же самых молекулах ДНК — технология, судя по всему, куда более близкая к практической реализации.

⇡#Обзор стоечного ИБП Ippon Innova RTB 3000

⇡#Сенатор США потребовал разъяснений по связям главы Intel с китайским бизнесом

⇡#Ещё одна миссия NASA под угрозой срыва — потеряна связь с зондом для изучения полярных щелей Земли
⇡#Всё уже эволюционировало до нас
Человечество тысячелетиями обнаруживало всё новые способы фиксации данных — на камне, дереве, глиняных табличках, папирусе и т. д., — пока не пришло к полупроводниковым и магнитным цифровым накопителям. Однако, строго говоря, что есть сама эволюция — носителем переходящего звания венца которой, если судить по наличию неплохой способности к абстрактному мышлению, как раз и можно считать человека, — как не непрерывное преобразование накопленной прежними поколениями информации ради наилучшего приспособления (к среде своего обитания) поколений настоящих и, в идеале, будущих? А раз биологическая информация между поколениями передаётся — с тем, чтобы где-то повториться и воспроизвестись, а где-то и измениться, — у неё должны быть материальные носители. Ещё в середине прошлого века появились неопровержимые доказательства, что такими носителями практически для всего живого на нынешнем этапе развития жизни выступают молекулы ДНК (в прошлом, вполне вероятно, существовал «мир РНК» — более простых биополимеров, способных к самовоспроизводству и выполняющих теперь в основном служебные функции; см. «центральная догма молекулярной биологии»). Как раз в те же годы знаменитый Ричард Фейнман (Richard P. Feynman) в своей известной и где-то даже провидческой лекции «Там, внизу, достаточно места» (Plenty of Room at the Bottom) предположил, что природное хранилище генетической информации можно использовать для записи каких угодно данных.
В принципе — оставляя пока в стороне вопрос о технической организации процедур чтения и записи — хранить данные в длинной цепочке дезоксирибонуклеиновой кислоты чрезвычайно просто. Каждый экземпляр ДНК представляет собой закреплённую на сахаро-фосфатном остове (том самом двуспиральном каркасе, что знаком каждому по популярным изображениям этой молекулы) последовательность нуклеотидов (азотистых оснований) четырёх разновидностей: аденин (A), тимин (T), гуанин (G) и циазин (C). Особая структура такой молекулы — двойная спираль, нуклеотиды обеих веток которой попарно соединены водородными связями, — обеспечивает её высочайшую стабильность: при температуре +9 °С отдельная ДНК — вне живой клетки — сохраняется не менее 2 тыс. лет, а оценка сохранности такой молекулы при −18 °С составляет уже 2 млн лет, что заведомо превосходит гарантированный срок работоспособности любого магнитного или полупроводникового носителя данных.
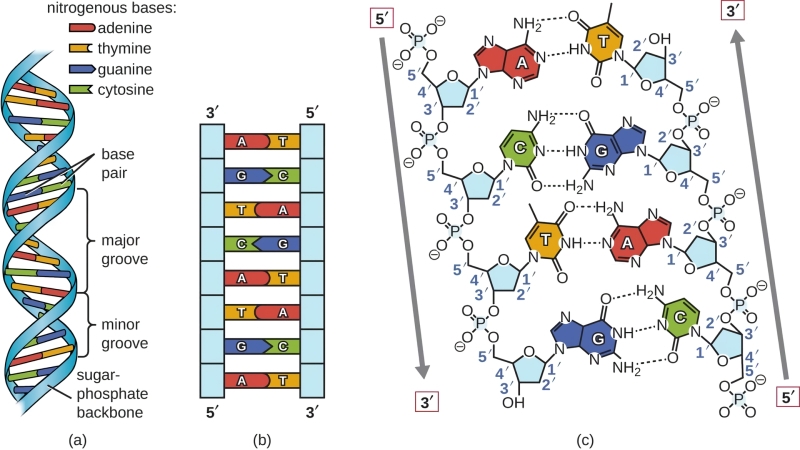
И хотя отдельная молекула — объект буквально наноразмерный (диаметр двойной спирали — 2 нм, расстояние между соседними парами нуклеотидов вдоль сахаро-фосфатных нитей — 0,34 нм), с чисто технической точки зрения она отлично подходит для достаточно надёжного сохранения данных. Ну ещё бы — такой её сделала продолжавшаяся миллиарды лет биологическая эволюция! Скажем, двунитчатая конструкция ДНК не только упрощает формирование пары идентичных копий сохраняемой в этой молекуле генетической информации (с прицелом на размножение), но и уберегает такую информацию от повреждений, например высокоэнергичными ультрафиолетовыми фотонами, точнее, позволяет специально для того развившимся внутриклеточным механизмам обнаруживать повреждённые участки на одной из нитей, вырезáть их и затем достраивать на освободившемся участке нужный фрагмент по комплементарной цепочке нуклеотидов на второй нити.
Бесспорно, как носители информации молекулы ДНК, скорее всего, будут храниться и эксплуатироваться для чтения и записи обособленно, за пределами клеточных стенок — в виде водного, к примеру, раствора, как в эксперименте Адлемана, — но работать с ними придётся как раз химическими методами; аналогичными тем, что действуют в живых клетках. Благо методы эти по сути незаменимы: если водородные связи между нуклеотидами в комплементарных нитях сравнительно хрупки — разрушаются при нагреве ещё до кипячения, например, — то азотистые основания в пределах каждой нити сцеплены настолько прочно, что разъединить их можно либо очень сильной кислотой, либо с применением особых ферментов, химерных нуклеаз, специально сконструированных для подобных операций. Грубо говоря, нуклеаза — составленное из двух структурных единиц белковое соединение, способное сперва избирательно связываться с определёнными последовательностями нуклеотидов в ДНК-мишени, а затем катализировать её расщепление («разрезать молекулу») на данном участке. В начале 2010-х был предложен ещё более эффективный метод генетической инженерии, CRISPR/Cas, где для распознавания целевого участка применяют не белки, а короткие молекулы РНК, но сам принцип «обнаружь — разрежь» так и продолжает полагаться на биологические механизмы.
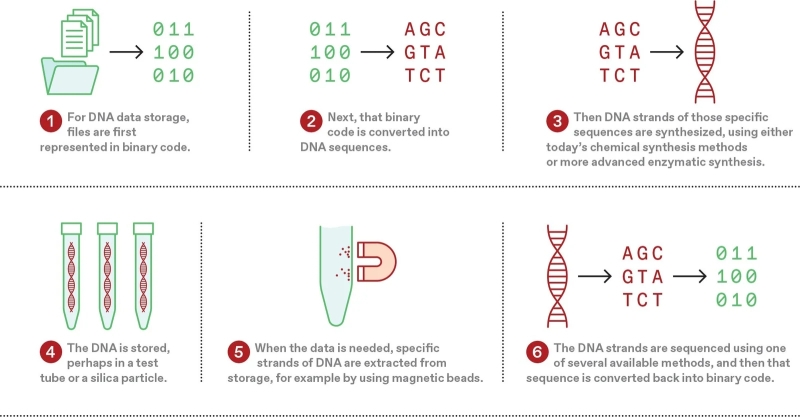
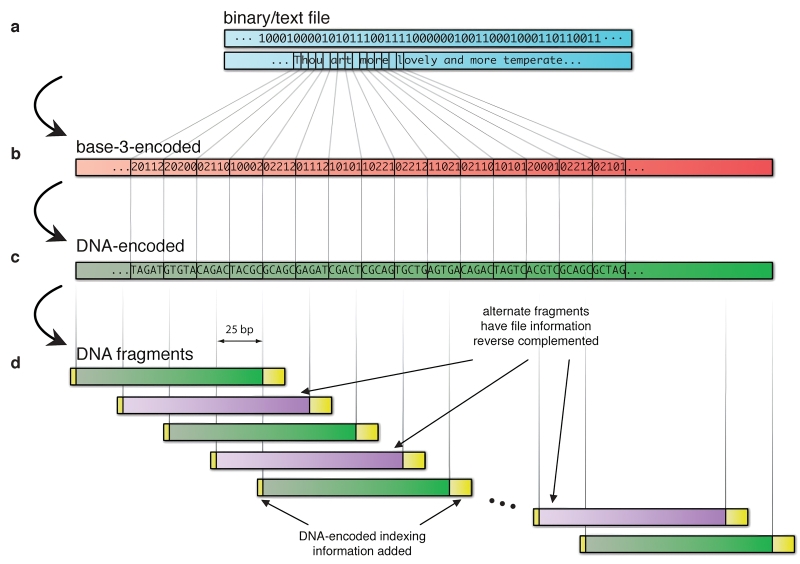
К настоящему времени очень неплохо отработаны, буквально в промышленных масштабах, технологии секвенирования ДНК, т. е. прочтения «записанной» в ней последовательности нуклеотидов, а также её синтеза — обратной задачи организации разрозненных азотистых оснований, которые можно рассматривать как элементарные единицы кодируемых данных, в прочную и долговечную молекулу. Ключевой этап — запись данных: информацию, предназначенную для хранения в ДНК, переводят из двоичного кода в назначенную последовательность нуклеотидов (где A в первом приближении может кодировать «00», T — «01» и т. д.), а затем сшивают эти нуклеотиды должным образом. Технологически процесс сшивания напоминает, скорее, последовательное наложение молекул одна на другую в процессе многоступенчатого химического синтеза, и исходно он мог занимать месяцы для нескольких десятков нуклеотидов, но над его ускорением и упрощением непрерывно работают.
Ещё в 2016 г. исследователи из Университета Вашингтона совместно с коллегами из Microsoft сообщили, что им удалось закодировать 35 разнородных файлов протяжённостью от 29 Кбайт до 44 Мбайт суммарным объёмом более чем в 200 Мбайт с использованием примерно 13 млн олигонуклеотидов — коротких, в 200 или менее азотистых оснований, фрагментов ДНК. Да не просто закодировать, а предложить способ последующего поиска и выборочного считывания записанных на молекулярном уровне данных. Достижение это примечательно как раз тем, что, хотя и ранее в лабораториях удавалось и кодировать информацию в искусственным образом компонуемых ДНК, и извлекать её, впервые на практике был продемонстрирован сравнительно быстрый и точный метод организации произвольного доступа к столь внушительному объёму сохранённых на молекулярном уровне данных.

⇡#Слишком много данных
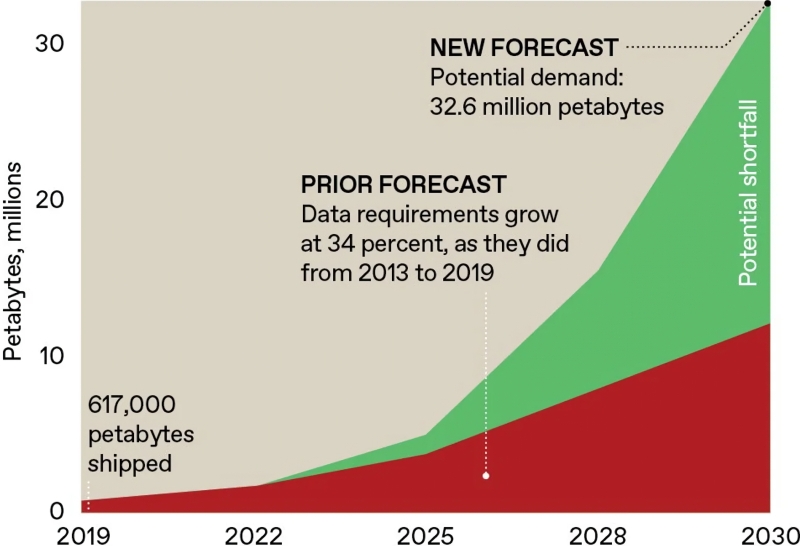
Почему так важно обеспечить возможность произвольно обращаться к молекулярным средствам хранения для выборки нужных файлов — ведь, к примеру, магнитные ленты, до сих пор применяемые для архивации, всех устраивают именно как устройства последовательного доступа? Если формально подсчитать, какое количество единичных нуклеотидов поместится в 1 мм3 при нормальных условиях (с некоторым допуском на то, что они будут располагаться не вплотную, а образуя классические двойные спирали), получим весьма воодушевляющий теоретический верхний предел плотности записи информации в ДНК — около 1 Эбайт на этот объём, или, в пересчёте на массу, 215 Пбайт в каждом грамме (при условии двухбитового кодирования без избыточности, правда, — что, как мы увидим позднее, чересчур оптимистично было бы допускать). Потому, если ограничиться просто декодированием всей записываемой на эти макромолекулы информации за один присест — а технически сделать это проще всего, ибо, как уже было сказано, секвенировать ДНК сегодня умеют сравнительно быстро и дёшево, — придётся записывать расшифровываемую информацию на некий внешний по отношению к пробирке с «растворёнными данными» носитель, а затем уже искать нужные файлы там. Такой подход лишает смысла само применение макромолекул в качестве независимых накопителей, основное преимущество которых — непревзойдённая плотность хранения. Не следует также забывать, что как синтез ДНК, так и её секвенирование — операции с чрезвычайно (по меркам полупроводниковых фон-неймановских вычислителей) высоким уровнем ошибок, а следовательно, необходимо предусматривать некие механизмы их компенсации/исправления, хотя бы банальные контрольные суммы и/или дублирование (а лучше страивание) последовательностей данных.
Предложенный исследователями в 2016 г. метод эффективно — хотя, возможно, и не совсем изящно — обошёл это ограничение посредством использования олигонуклеотидных праймеров (очень коротких — порядка 20 азотистых оснований — фрагментов одиночной ДНК-нити) в сочетании с полимеразной цепной реакцией (ПЦР). Реакция эта, сегодня чрезвычайно широко применяемая в медицинских исследованиях, позволяет создавать — с довольно скромными трудозатратами — копии заданного фрагмента имеющейся в наличии ДНК. История метода ПЦР тянется аж с 1957 г.; в данном же случае важно, что его применение повышает концентрацию определённой последовательности нуклеотидов — если та исходно имеется в пробе — сразу на несколько порядков, что делает обнаружение искомой последовательности делом практически тривиальным. Собственно, как раз благодаря ПЦР сегодня так быстро и надёжно диагностируются некоторые инфекционные заболевания (по «молекулярным отпечаткам» генетического материала провоцирующих их микроорганизмов), подтверждается/опровергается принадлежность подозреваемому обнаруженных на месте преступления биологических материалов и т. д.
По сути, новаторство исследователей из Университета Вашингтона и Microsoft заключалось в том, что они — на предваряющем кодирование записываемых в ДНК данных этапе — разработали обширную библиотеку целевых праймеров, которые затем цепляли к обоим концам каждой из последовательностей нуклеотидов, содержащих определённый фрагмент информации. Итоговая большая ДНК формировалась, таким образом, из составленных встык помеченных фрагментов, и на этапе поиска случайный доступ реализовывался предельно естественным образом: если известно, какие именно цепочки азотистых оснований помечены определёнными праймерами, ПЦР-реакция даёт возможность повысить концентрацию именно этих цепочек в растворе до такого уровня, что последующее секвенирование производится довольно быстро и с минимумом ошибок (для коррекции которых, в свою очередь, исследователями были разработаны подходящие как раз для данного метода алгоритмы).
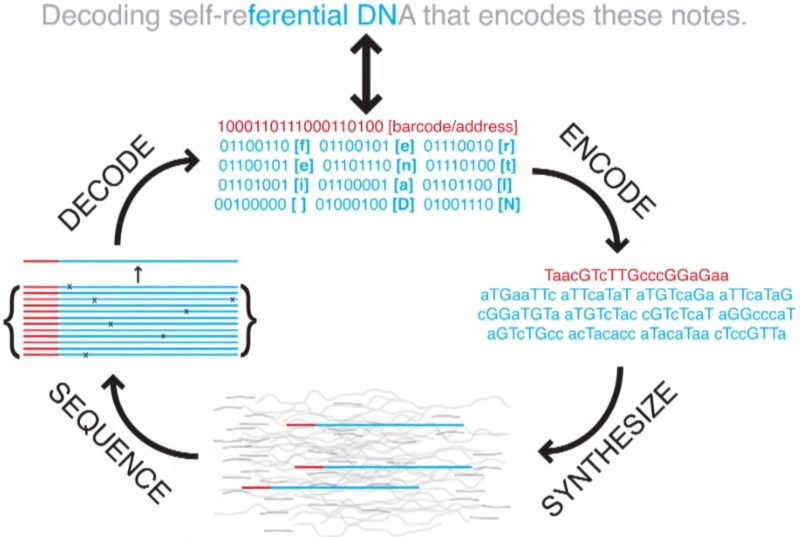
Каким же, собственно, образом производится первичное молекулярное кодирование данных — создание базовых синтетических фрагментов, которые затем оформляются с торцов каталогизированными праймерами и идут на формирование итоговой общей ДНК? Здесь на помощь приходит молекулярная химия: отдельные нуклеотиды соединяются между собой, формируя полимерную цепочку, посредством фосфодиэфирных связей последовательно, на протяжении ряда стадий. Сперва соединяют два мономера (отдельных нуклеотида; скажем, A и G) — в макроскопическом объёме, разумеется; в ходе химической реакции с активацией фосфатной группы, а не условным пинцетом под не менее условным микроскопом, — затем к раствору полученного димера добавляют следующий необходимый нуклеотид (допустим, С), получая таким образом тример, и так далее — пока не будет сформирована вся цепочка требуемой конфигурации. В процессе необходимо тщательно контролировать однородность получаемых продуктов, вводя защитные реагенты для блокирования нежелательных реакций. К настоящему времени известны и более прогрессивные (скоростные, дешёвые и обеспечивающие высокий уровень чистоты продукта на каждой стадии) методы с использованием твердофазного амидофосфитного синтеза в автоматических синтезаторах, так что создание коротких последовательностей нуклеотидов серьёзной проблемой можно уже не считать. Ещё в 2018 г. компании вроде GenScript Biotech и Integrated DNA Technologies предоставляли всем желающим услугу синтеза двухнитевых ДНК-фрагментов протяжённостью до нескольких сотен пар азотистых оснований по цене всего 10-40 центов за каждую пару; сегодня это обходится ощутимо дешевле.
Само собой, не всё так просто: даже с учётом весьма высокого качества каждого амидофосфитного синтеза по отдельности — в 99% случаев к формируемой цепочке нуклеотидов на очередном этапе присоединяется именно тот, что необходим, — в итоге процедуры, требующей последовательного проведения сотни таких реакций, лишь 37% итогового продукта в точности соответствуют требованиям заказчика. Собственно, это одна из важнейших причин, по которым описанным методом — вполне себя оправдывающим для коммерческих приложений, уверенно автоматизированным — удаётся получать лишь олигонуклеотиды, т. е. сравнительно (с природными ДНК) короткие молекулы: едва ли не экспоненциальный характер накопления ошибок обессмысливает синтез длинных цепочек оснований. Тем не менее сборка из размеченных тщательно подобранными праймерами олигонуклеотидов молекул большой протяжённости вполне себя оправдывает — однако здесь уже необходимо привлекать не столько химические, сколько математические и кибернетические компетенции.
⇡#Дела весьма грядущих дней
Для начала следует по максимуму обезопасить сохраняемую на ДНК информацию от появления и накопления ошибок, которые неизбежно будут возникать в ходе химических реакций, протекающих в пусть и небольших (вплоть до микролитров), но всё же макроскопических — по сравнению с единичной молекулой — объёмах. В начале 2010-х сразу две группы исследователей, из Гарварда и из британского Европейского института биоинформатики, предложили довольно сложно организованную, но, по их заверениям, крайне надёжную схему кодирования информации для ДНК-накопителей. Прежде всего, от бинарного представления данных рекомендовано было перейти к троичной системе (с основанием не «0» и «1», а «0», «1» и «2»). Почему именно троичной? Да потому, что длинные последовательности однотипных нуклеотидов (AAAAAA, GGGG, да даже банально CC) в ходе секвенирования часто воспроизводятся с ошибками, и потому имеет смысл уже на самом раннем этапе обеспечить как можно более частое чередование оснований в их цепочке — в идеале не допуская даже простых повторов. По этой причине между «0», «1» и «2» троичной системы кодировки и четырьмя типами оснований — A, T, G, C — ДНК-носителя в предложенной схеме нет однозначного соответствия. Вместо того действует допускающее чёткую алгоритмизацию, но более сложное правило: «если в кодировке по основанию 3 следуют подряд два одинаковых символа, нуклеотиды для их обозначения берутся разные, с определённым сдвигом». Пример: пусть некая литера в троичной кодировке представлена как 20112, тогда в переложении на нуклеотиды ей будет соответствовать последовательность TAGAT — здесь первая из пары соседних единиц представлена как G, а вторая уже как A, хотя буквально только что та же самая A обозначала «0». Вполне возможно оказывается составить таблицу взаимно однозначных соответствий таких замен именно в случае кодировки по основанию 3 с применением 4 нуклеотидных «букв» — и затем пользоваться этой таблицей как при записи информации в ДНК, так и на этапе считывания.
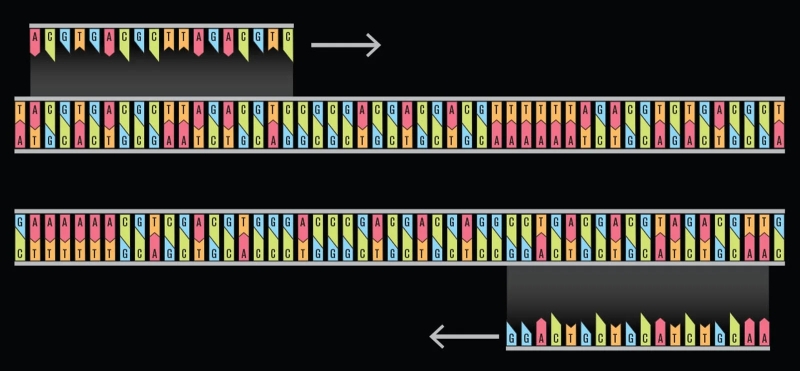
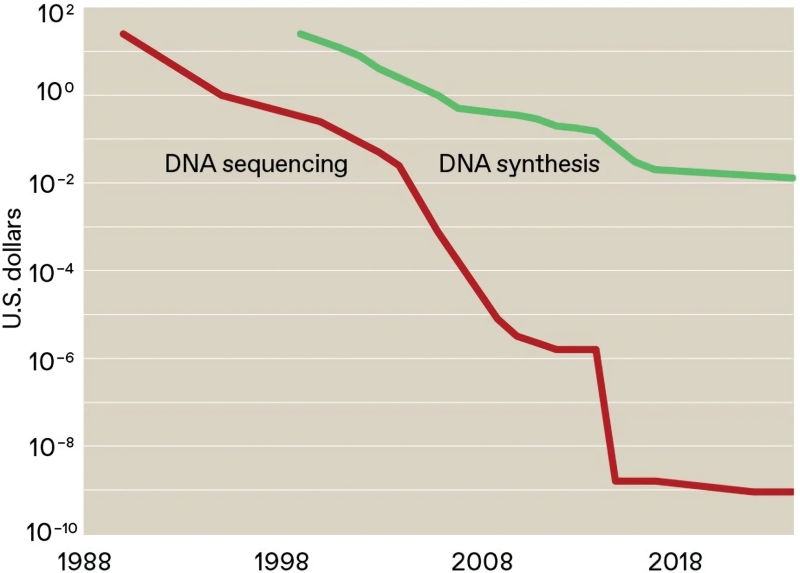
Второй важный момент: данные в ДНК записываются с неимоверной, по меркам полупроводниковых либо магнитных носителей, избыточностью — 75%. То бишь если итоговая длинная макромолекула составлена из элементарных блоков по 100 нуклеотидов (в некотором смысле это аналоги секторов на поверхности магнитного жёсткого диска), то в каждом последующем блоке только 25 последних пар оснований окажутся оригинальными; первые же 75 будут в точности повторять концевые 75 пар из предыдущего блока. Причина очевидна: хотя молекулярные логические вентили для операций с олигонуклеотидами уже предложены, их добавление в систему, предназначенную только для хранения данных, несоразмерно усложнит её и удорожит. А отсутствие вычислительной логики делает невозможным применение одного из наиболее эффективных средств обнаружения/исправления ошибок — генерации и регулярной сверки контрольных сумм. Так что вящая избыточность троично-четверичного представления данных в ДНК (троичная система условного представления символов; четыре нуклеотида для её физического воплощения) остаётся в такой ситуации единственно надёжной гарантией от появления значимого числа ошибок в ходе как записи, так и чтения. Понятно, что дешевле хранение информации на макромолекулах от этого не становится: в 2013 г., когда американские и британские исследователи предложили описанный метод, средняя стоимость записи 1 Мбайт информации в ДНК (с разбиением её на короткие блоки, с последовательным синтезом соответствующих олигонуклеотидов в специализированных лабораториях и сшивкой их затем в единую молекулу) достигала 12 тыс. долл.; считывания того же самого 1 Мбайт — 220 долл. Сегодня эти суммы на порядки ниже, и как раз потому в наши дни ДНК в качестве долговременного носителя архивных данных привлекает всё больше практического внимания.
Впрочем, как это сплошь и рядом бывает что в микроэлектронике, что в молекулярной химии, дальше начинаются всевозможные тонкости (но они-то и делают что научный, что инженерный поиск подлинно захватывающим!). Казалось бы, вот она, удачная и простая схема организации данных в ДНК: как дорожка записи на магнитном диске размечена на сектора, так и длинная, несущая информацию макромолекула составлена из олигонуклеотидных блоков, каждый из которых помечен однозначно идентифицирующими его праймерами. Для извлечения же данных остаётся лишь свериться с каталогом праймеров (разумеется, расположенным на внешнем носителе, скорее всего, как раз классическом — полупроводниковом или магнитном), подготовить соответствующие им полимеразы, произвести ПЦР и получить на выходе тот участок исходной ДНК, на котором записана требуемая информация.
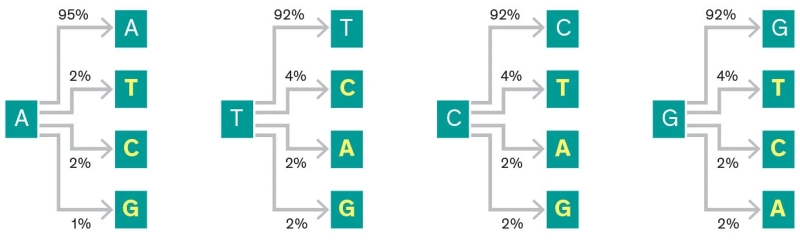
Увы, на практике реализация этой вполне прозрачной схемы сопряжена с немалыми трудностями: праймеры необходимо изначально подбирать так, чтобы они связывались только с соответствующими олигонуклеотидами, и не с какими иными из образующих общую итоговую ДНК, — а чем та длиннее, тем, ясное дело, более трудоёмкой и нерядовой становится эта задача. Далее, между формирующими ДНК нуклеотидами есть небольшие различия: формально однотипные водородные связи между парами, что составляют «ступеньки» всем на вид знакомой спиральной лесенки, в случае соединения G—C чуть сильнее, чем для A—T. А это, в свою очередь, означает, что, если некий праймер будет содержать слишком много оснований A и T, его связь с соответствующим участком ДНК напротив окажется слабее, чем у прочих, то есть операции с ним в ходе ПЦР будут проводиться с более сжатым таймингом, что практически наверняка приведёт к появлению дополнительных ошибок в ходе секвенирования.
Мало того; различные секвенсоры (автоматизированные аппараты для, собственно, секвенирования геномов) от разных разработчиков своеобразны в том смысле, что склонны чаще допускать определённые ошибки: одни порой (и чем длиннее молекула, тем чаще) подменяют основания — вместо A ставят на эту позицию, допустим, C; другие случайно «теряют» — не включают в итоговую расшифровку ДНК — целые олигонуклеотидные блоки; у третьих в процессе декодирования из цепочки оснований бесследно исчезают отдельные пары и т. п. По счастью, зная, каким именно ошибкам подвержен данный конкретный секвенсор, можно ещё на уровне троично-четверичного кодирования данных, чисто математически, предусмотреть дополнительные механизмы обнаружения и компенсации его будущих промахов. Но это, в свою очередь, лишает универсальности ДНК как носитель данных! Выходит, для расшифровки сохранённой с расчётом на воспроизведение на конкретной установке информации необходимо будет применять только эту самую установку; и даже замена её прямой наследницей — у которой некоторые специфические ошибки будут проявляться реже — может попросту ухудшить, а вовсе не повысить качество воспроизведения данных. Спрашивается, стоит ли вообще городить огород с ДНК-хранением, рассчитывая передавать информацию в макромолекулах через века и тысячелетия, если нет даже твёрдой гарантии, что отдалённые наши потомки (или какие-нибудь залётные рептилоиды с Нибиру) смогут в принципе разобраться, где именно эти доисторические затейники разместили в этой конкретной цепочке нуклеотидов содержательные данные — и каким хитроумным образом их закодировали?
В итоге выясняется, что создание ДНК-накопителей требует теснейшей кооперации специалистов в областях синтетической биологии и криптографии (точнее, теории кодирования данных — поистине обширной области прикладной математики) — чтобы предельно эффективно снимать проблемы, порождаемые несовершенством нынешних технологий органического синтеза и секвенирования. Впрочем, схожего результата можно, судя по всему, достичь, развивая эти самые технологии нетривиальными путями — с переходом, в частности, от традиционного «мокрого» синтеза/секвенирования в макроскопическом объёме жидкости к построению упорядоченных полимерных каркасов, в волокнистые структуры которых вплетаются отдельные молекулы ДНК. Здесь вместо того, чтобы городить огород с ПЦР, исследователи из Университета Северной Каролины и Университета Джонса Хопкинса в 2024 г. предложили вернуться к природному методу считывания, копирования и перезаписи ДНК, а именно — с использованием специализированных для этих целей молекул РНК (что порождает, ясное дело, массу иных проблем, но зато снимает немалый объём ныне актуальных). Другой потенциально прорывной метод развивают в последние годы в КНР: он предусматривает использование в качестве непосредственных носителей данных не самих нуклеотидов, образующих гены, а «эпибитов», которые формируются на основе нуклеотидов в процессе метилирования (играющем, кстати, немалую роль и в природной, биологической области функционирования ДНК, — см. «эпигенетика»). Поскольку в данном случае в принципе не требуется ничего вырезать и вставлять — сама двухнитевая цепочка оснований сохраняется в неизменности, — процессы чтения и записи значительно ускоряются, достигая умопомрачительных для биомолекулярных хранилищ данных величин в 40 бит/с (прототип Университета Вашингтона и Microsoft 2018 г., о котором мы упоминали, обрабатывал 5 байт на протяжении 21 часа).
Наконец, уже в 2025 г. у исследователей из Техасского университета в Остине родилась идея синтезировать конструктивно схожие с ДНК молекулы из «позиционно-определяемых полимеров» (sequence-defined polymers). Бесспорное достоинство такого метода — в возможности не ограничивать себя на самом базовом уровне кодировки: вместо четырёх «природных букв» G, C, A и T техасцы сходу создали 256 элементарных компонентов с различными электрохимическими свойствами, что позволило заметно повысить точность их идентификации в растворе — а следовательно, ускорить операции чтения/записи с одновременным снижением частоты проявления ошибок. И даже тот факт, что экспериментальный синтетический полимер в процессе чтения разрушается, можно обратить на пользу дела — применяя такой подход, например, для обмена чувствительными данными с необходимостью гарантировать, что те не были скомпрометированы в процессе передачи.
Словом, пока что главным двигателем прогресса в области ДНК-накопителей продолжает оставаться обеспечиваемая ими невероятная — по меркам едва ли не всех прочих носителей данных — плотность хранения: все 120 Збайт, в которые на начало 2024 г. оценивался объём доступной через Интернет информации, теоретически должны уместиться всего в 1 см3 этих молекул (уже не в составе раствора, разумеется, а просто уложенных плотненько одна к одной, — и если оставить в стороне вопрос о том, сколько времени потребуется на подготовку данных к кодированию и собственно на формирование этого кубика). В отличие от магнитных лент, ДНК при должным образом организованных условиях хранения не деградирует по меньшей мере столетиями, не требуя притом ни электроэнергии для поддержания своего состояния, ни периодической проверки работоспособности носителя с возможной последующей перезаписью, — для архивации огромных массивов данных (да, мы смотрим на вас, тренировочные БД для обучения всё более и более масштабных ИИ) это едва ли не идеальный вариант.
В 2020 г. рядом исследовательских университетов, коммерческих и финансируемых из бюджетов лабораторий был основан DNA Data Storage Alliance — международный консорциум, ставящий своей целью ускорить преодоление тех преград, что ныне задерживают развитие и внедрение ДНК-хранилищ. А это в первую голову — непозволительно (с точки зрения повседневных задач архивации) низкая скорость, которую, чтобы довести до достигаемой современными магнитными лентами 2 Гбит/с, придётся каким-то образом увеличить от нынешних невеликих значений — около 300 тыс. нуклеотидов в секунду; именно нуклеотидов; полезных данных «нетто» там куда меньше из-за огромной избыточности записи — как минимум на четыре десятичных порядка. Цель, что и говорить, амбициозная — посмотрим, какими темпами будут двигаться к ней разработчики! По крайней мере, потребность в огромных хранилищах данных с минимальной, а лучше нулевой энергетической ёмкостью год от года становится всё острее, а значит, стимул к совершенствованию ДНК-накопителей имеется преизрядный.
Материалы по теме
- Учёные придумали хранить данные в пластиковом аналоге ДНК — это будет плотно и надёжно.
- Китайцы предложили навечно записывать данные в алмазах — плотность будет в 10 000 раз выше, чем на DVD.
- Разработана технология записи данных в существующую ДНК.
- Учёные создали основу для будущих ДНК-компьютеров, которые одновременно хранят и обрабатывают данные.
- Учёные облачили ДНК в искусственный янтарь — получилось сверхплотное и долговечное хранилище данных.
- Китайские учёные представили базу для создания универсальных компьютеров на ДНК.
- Биологическая совместимость.
